Cette page est expérimentale, son but est de vous faire travailler pas à pas en ayant un accès direct aux ressources de wikipedia et d'autres sites spécialisés ; et en vous permettant de faire des exercices et de rédiger des fiches de synthèse. Merci de vos retours et conseils sur le fond (erreurs à corriger) ou la forme (améliorer l'ergonomie, la lisibilité ...) ; je ferai ce que je peux ! 😉 J.D.
La chromatographie d'échange d'ion (IEX)
Principes théoriques
Principes théoriques
Rappel sur la charge des protéines
Les protéines sont constituées des 20 acides aminés assemblés
par le ribosome lors de la traduction et de quelques acides
aminés dérivés des 20 premiers par modification
post-traductionnelle (hydroxy-proline, hydroxy-lysine, ...).
Certains ont des chaînes latérales hydrophobes,
d'autres hydrophiles, et parmi ces derniers, certains ont des
chaînes latérales partiellement ionisées, donc chargées
Quels sont les acides aminés chargés ?
Sources wiki :
Rappels sur la force des acides et des bases : phases fixes chargées
Définition d'un acide : molécule capable de fournir un ou plusieurs protons (H+) à une base ;
remarques : :
- pour une base, c'est l'inverse !
- les groupements chimiques échangeurs d'ions sont très souvent des acides ou des bases ou leurs sels …
- H2O est à la fois un acide et
une base ;
écrire les deux équations ;
Proposition de réponse
- acide : H2O ⇆ H+ + HO-
- wiki sur les acides
- base : H2O + H+ ⇆ H3O+
- wiki sur les bases
Acides et bases fortes
AHn → An- + n.H+
de même
BOH → Bn+ + n.OH-
-
Les acides forts sont par exemple les acides chlorhydrique (HCl), sulfurique (H2SO4) qui sont entièrement ionisés dans l'eau et donnent respectivement : une mole de protons (hydronium = proton + H2O = H3O+)
par mole d'HCl ; et deux moles pour l'acide sulfurique.
Leur réaction est brutale, totale et irréversible lorsqu'ils sont dissouts dans l'eau (attention les projections !) ; autrement dit, un acide fort donne tous ses protons à l'eau accompagné d'un ion spectateur stable (pour nos deux acides ce sera le chlore et le sulfate). Un acide ou une base forte ont dans leur équation réactionnelle une flèche à sens unique ! Les bases fortes utilisées en biologie sont souvent des oxydes de métaux alcalins :
- CaO (chaux vive) qui donne après hydratation la chaux éteinte Ca(OH)2 ; elle n'est utilisée que pour mettre en évidence le gaz carbonique dans l'air …
- la potasse ou KOH est utilisée pour saponifier les corps gras de type diacyl (phospholipides) ou triacyl glycérol (les triglycérides : huiles et graisses alimentaires) ;
- c'est surtout la soude NaOH qui est la plus utilisée pour alcaliniser les milieux de culture et pour ajuster le pH des tampons.
Acides et bases faibles
Les acides et bases faibles sont des molécules dont l'acidité ou l'alcalinité n'est pas complète : dans l'eau elles sont sous forme d'un mélange entre leur forme acide et leur forme basique
La formule du pKa permet de le montrer
Le pKa est un pH particulier à chaque acide ou base organique qui exprime le pH pour lequel il y a autant de formes acides (AH ou BH+) que de forme basique (A- ou B).En biologie, la plupart des acides faibles contiennent des groupements carboxyliques (-COOH) ou phosphoriques (-o-P(OH)2) et les bases sont essentiellement des composés de l'azote (-NH2).
Mise en oeuvre technologique
Mise en oeuvre technologique
Les différentes résines échangeuses d'ions
Les échangeuses d'anions
Les échangeuses d'anions fortes
Elles sont dotées de groupements chimiques stables dont
la charge + ne varie pas en fonction du pH ; la plus
commune est de type Q (pour Ammonium Quaternaire) ; en effet
un ammonium quaternaire n'est lié directement à aucun proton
échangeable, ses quatres partenaires sont souvent organiques
; par exemple : 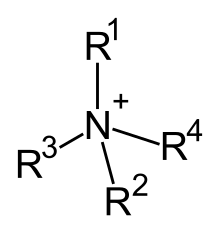
Les résines Q sont utilisées par exemple pour purifier l'ADN, l'ARN et les protéines acides ; l'élution se fait préférentiellement par compétition.
Proposer une composition de tampon d'élution dans ce cas.
proposition de réponse
L'élution se fait avec un tampon riche en cathions (NaCl concentré tout simplement).Les échangeuses d'anions faibles
Ce sont des amines primaire, secondaire ou tertiaires, donc des acides faibles. Par conséquent leur charge varie en fonction du pH et donc leur capacité de rétention.
Est-ce que leur capacité de rétention augmente si le pH augmente ?
Proposition de réponse
NON ! : à pH très acide la charge des bases faible est maximale car elles sont majoritairement sous leur forme ammonium positif ; donc quand le pH augmente, leur charge diminue et leur capacité d'échange aussi !
Une des résines de ce type les plus utilisées, est le
DEAE cellulose

Le DEAE est il un ammonium primaire, secondaire ou tertiaire ?
Réponse
Tertiaire puisqu'il y a trois molécules organiques liées à l'azote.
Les échangeuses de cathions
Ce sont des résines (polymères) chargées négativement.
Les échangeuses de cathions fortes
Pour la plupart elles sont contiennent des groupements sulfates (ion spectateur de l'acide fort sulfurique), dont la charge est permanente et ne dépend pas du pH !
Les échangeuses de cathions faibles
Ces résines sont souvent constituées de groupements acides carboxyliques liés covalemment à une résine : carboxy-méthyl cellulose par exemple.